一、 DNA甲基化
1.什么是DNA甲基化?
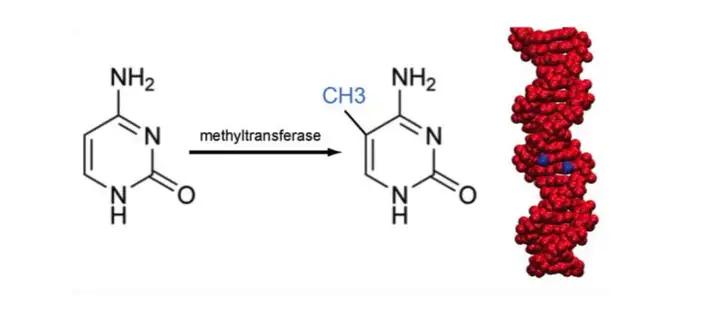
DNA甲基化是在DNA甲基化转移酶(Dnmt)的作用下将甲基选择性地添加到胞嘧啶上形成5-甲基胞嘧啶(5-mC)的过程。
2.DNA甲基化与基因表达调控
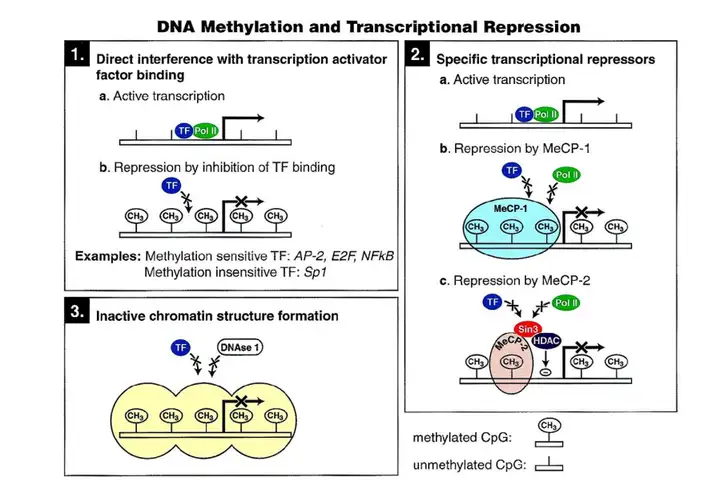
DNA复制后胞嘧啶的甲基化会改变DNA的构象,使DNA的大沟无法与DNA结合蛋白正常结合,从而使这些非编码区长期保持无表达活性的状态。而有转录活性的基因可利用非甲基化的启动子来进行转录表达。
3.DNA甲基化主要发生在CpG位点
CpG位点(CpG sites)是指DNA的某个区域,其上的碱基序列以胞嘧啶接着鸟嘌呤出现。“CpG”是“C—磷酸—G”的缩写。 CpG位点在人类基因组上并非随机分布,在人类基因组上的频率为1%。
• DNA甲基化主要发生在CpG位点。在哺乳动物中,70%到80%的CpG位点的胞嘧啶是甲基化的。
• CpG岛是一个富含CpG位点的区域,通常定义为:一个长度至少为200bp的片段
,其GC含量高于60%,主要位于基因的启动子区(70%的基因)。
二、全基因组甲基化研究策略
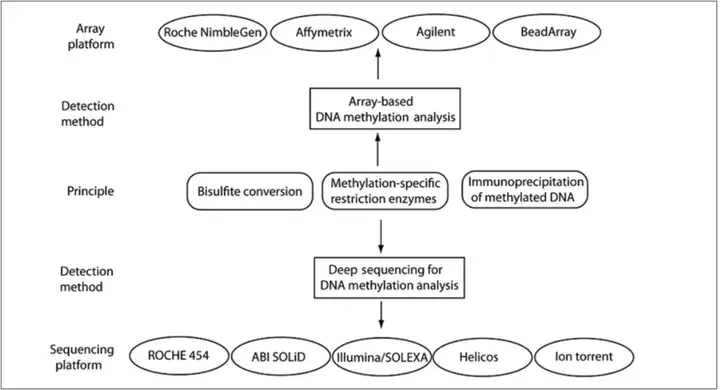
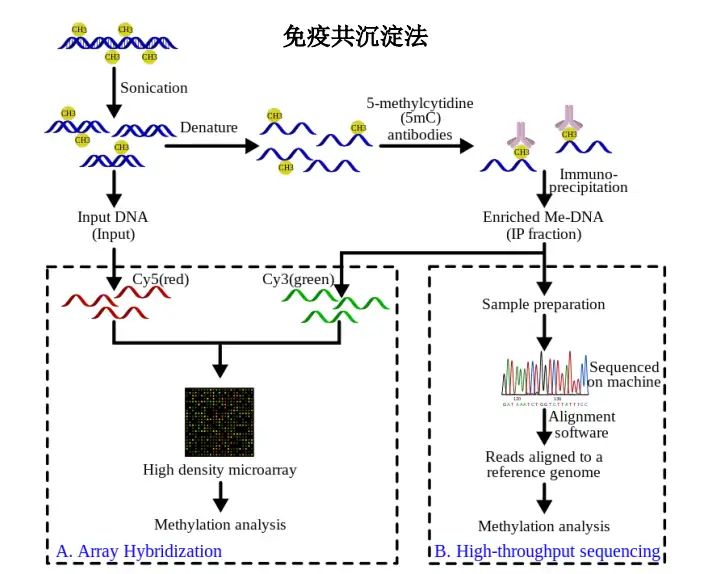
优点:通量高 ,全基因组范围;单碱基分辨率。
缺点:成本高,不适于大样本研究分析;不适于特异位点、基因或区段研究。
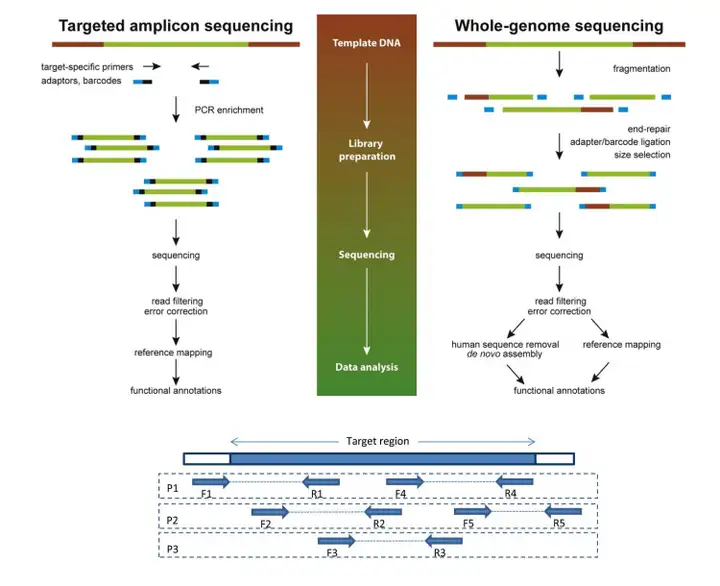
三、特异位点甲基化研究策略
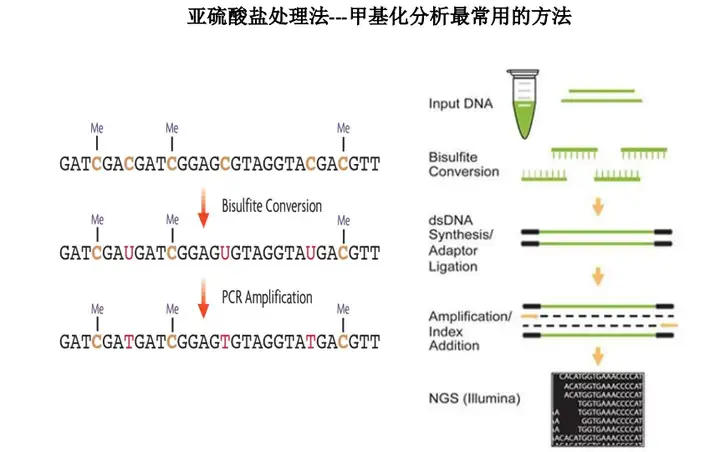
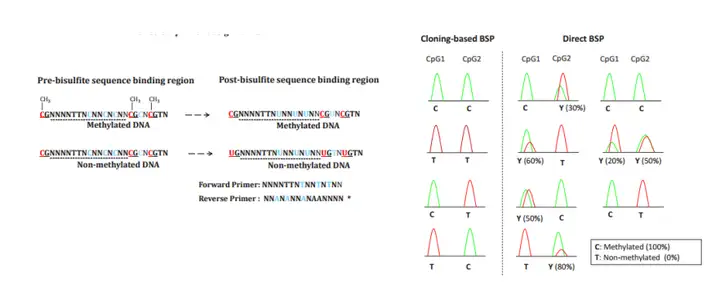
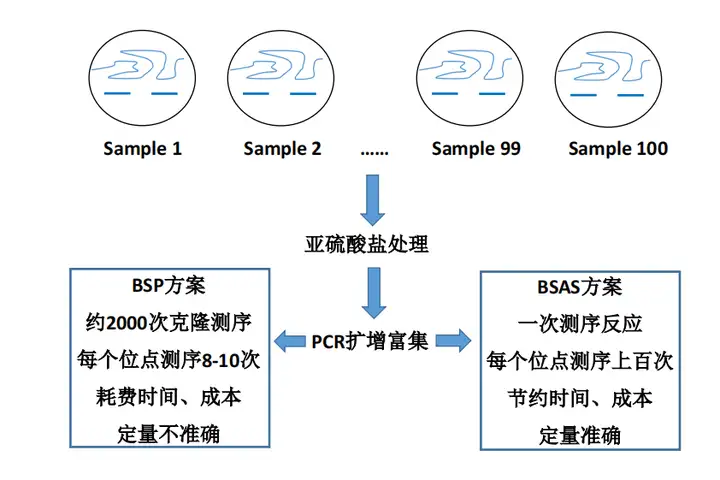
Bisulfite sequencing PCR (BSP): 基因组DNA经亚硫酸氢盐处理后,设计BSP引物扩增目的片段,最后对PCR产物进行克隆并选取8-10个转化子序列,即可确定所研究的CpG区域的甲基化频率。
Methylation specific PCR (MSP) :首先用亚硫酸氢钠修饰处理基因组DNA,然后针对甲基化和非甲基化引物进行PCR扩增,确定与引物互补的DNA序列的甲基化状态。
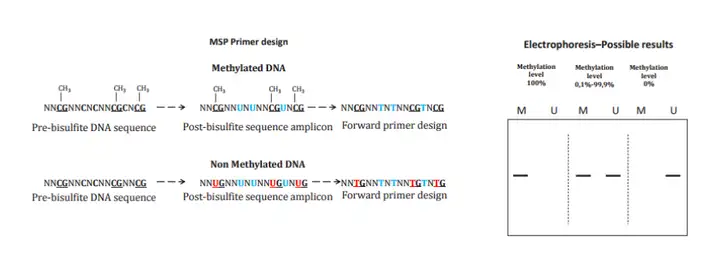
缺点:通量太小、无法精确定量
翼和方法:
Bisulfite Amplicon Sequencing (BSAS)
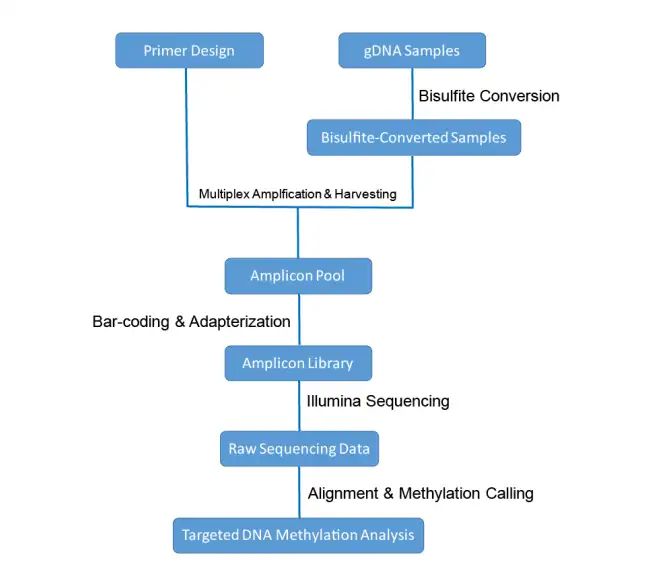
优势:BSAS 结合了亚硫酸盐转换、靶向扩增子高通量测序技术,可实现多区段、多位点的甲基化精确定量分析。
应用:1、 适用于感兴趣的目的片段的甲基化研究 ;
2、适用于在大样本中进一步确认全基因组甲基化研究挑选的阳性位点。
文献:1、 Masser et al. Epigenetics & Chromatin 2013, 6:33
2、Ashktorab et al. Epigenetics 2014, 9(4): 503-512
3、Beth et al. J CHILD PSYCHOL PSYC 2016,2(57):152-160
关键点-引物设计
尽量避免CpG位点,无法避免时,可设计兼并引物;
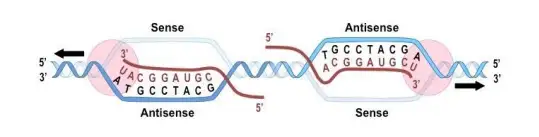
经亚硫酸盐处理后,两条DNA链不再互补,一般需要设计链特异性引物用于PCR扩增;
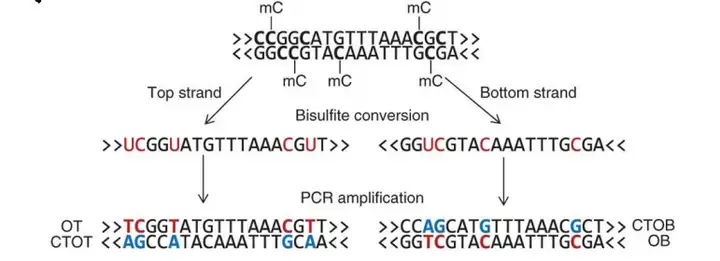
如果只选一条链进行研究,一般选择正义链。
关键点-转换效率
亚硫酸盐处理是一化学过程,可造成DNA的损伤,很难同时实现100%的转换效率和保持DNA的完整性,二者需要做出一定的平衡。
转换效率 = 未转换碱基读数/总碱基读数 【针对DNA序列中的C位点】
如何评估转换效率?
在植物中,叶绿体中不含甲基胞嘧啶 DNA,因此,可用叶绿体DNA序列信息作为内对照,评估转换效率。
在动物中,可通过在试验中加入未发生甲基化的外源DNA序列(通常为噬菌体DNA)来评估转换效率;此外,还可利用CpG位点以外的C位点信息来评估。
关键点-目的片段富集
采用巢式PCR,确保目的片段有效富集
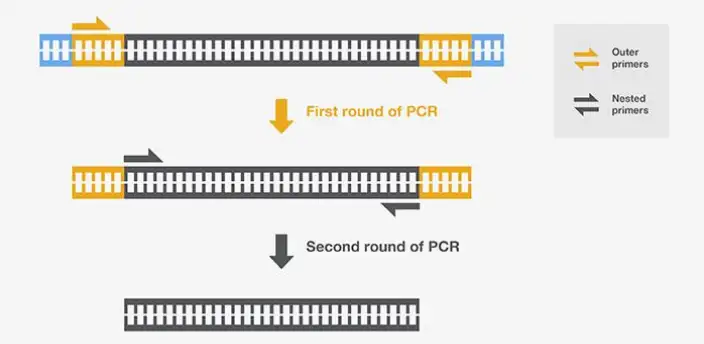
经亚硫酸盐处理后,DNA序列复杂度严重降低。应严格控制建库PCR的循环数,降低非特异性扩增。
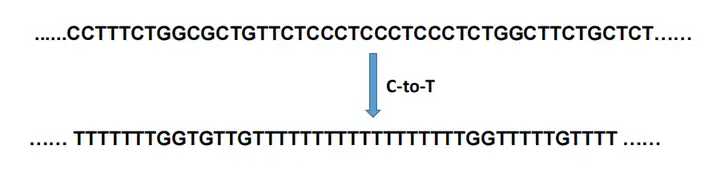
关键点-甲基化判断
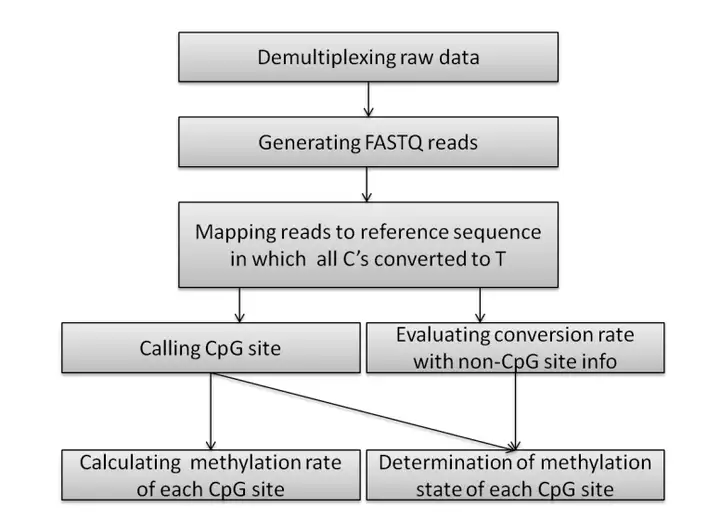
通过将参考序列中的C碱基全部替换为T碱基,然后将测序reads比对到转换后的参考序列上,针对每一个CpG位点,统计C和T碱基的读数(类似于SNP calling),来判断该位点的甲基化。
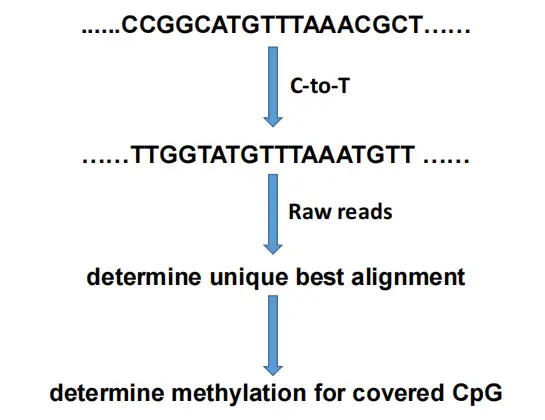
甲基化率:对于每一个CpG位点,甲基化率 = C碱基读数/该位点测序覆盖度。

甲基化状态:利用内对照得出的转换效率作为期望概率,通过二项分布统计,得到p值,判断每个CpG位点是否发生甲基化,多位点分析时,通常需要对p值进行矫正(错误发生率,FDR)。